王勇教授团队在深海微生物研究领域取得新进展
在浩瀚无垠的深海世界中,亿万微生物扮演着调控地球元素循环的重要角色。然而,我们采集到的样本,是否真的能代表微生物在深海原位的真实状态?近日,清华大学深圳国际研究生院王勇教授团队系统地评估了不同深海采样方法对深海活跃微生物的群落结构、核心代谢和环境响应基因表达的影响,为准确揭示深海微生物生态功能提供了技术路径。
在微生物生态学研究中,RNA是判断“谁在做什么”的核心分子。通过提取环境样本中的RNA,研究团队可以探究哪些基因正在表达,从而分析微生物的活性、功能和代谢路径。但RNA也极其“脆弱”——温度升高会促进其降解,压力变化和氧浓度变化会激活微生物的一系列应激反应,微生物转录组的整体结构可在几分钟内发生剧烈改变。然而,传统的Niskin瓶采样法却包含这些扰动:从深海采样后,水样需缓慢升至水面,期间温度从低于4°C升高至常温,压力由上百个大气压降至常压。这样的采样方式,虽然操作简便、应用广泛,却可能使样品中的微生物“发生表达漂移”,使研究人员观测到的是“扰动后的转录组”,而非原生态系统下的活跃状态。
为了避免传统采样过程中的转录扰动,王勇教授团队研发了两种深海原位采样技术,分别命名为MISNAC(Multiple In Situ Nucleic Acid Collection)和ISMIFF(In Situ Microbial Filtration and Fixation)。其中,MISNAC设备集成了深海自动抽滤、细胞裂解和RNA即时提取功能,能够在数分钟内完成海水样品的采集、处理和RNA稳定保存。整个过程无需人为干预,最大限度保持了微生物转录状态的原貌。而ISMIFF则采用原位抽滤与化学固定技术,通过深海现场注入RNAlater,将微生物细胞在过滤膜上即时固定DNA&RNA。此方法虽然不具备RNA即时提取能力,但仍可有效避免回收过程中的基因表达变化。相比之下,传统的Niskin瓶和仅进行原位过滤但不固定的ISMIFU(In Situ Microbial Filtration without Fixation)在RNA样本质量和转录保真度上存在短板。
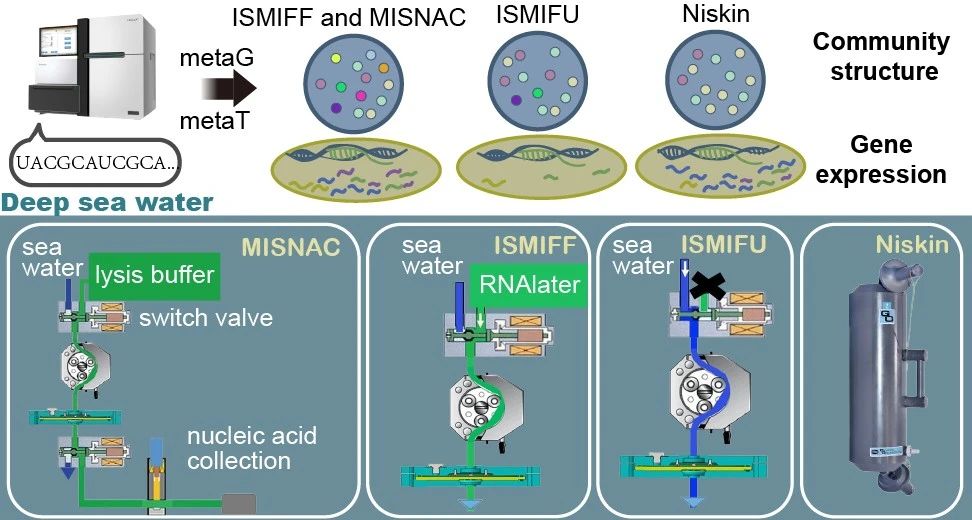
采用MISNAC、ISMIFF、ISMIFU以及Niskin采水瓶获取的深海微生物样品在转录组上表现出不同的微生物群落组成和基因表达水平。
为了验证不同采样方式对转录组结构的影响,研究团队选择在中国南海水深约1000米的站位开展对比实验。利用MISNAC、ISMIFF、ISMIFU和Niskin四种方法同步采集微生物样本,并通过高通量测序技术获取其宏基因组与宏转录组数据。不同采样方式获得的样品的转录组中微生物群落差异较大。原位样本中特有高表达的物种包括Nitrosopelagicus(氨氧化古菌)、SAR324、Marinimicrobia等深海特有类群,而Niskin样本则被Alteromonas、Oleibacter等可快速增殖的“扰动响应者”主导。这项研究从根本上指出了一个被长期忽视的问题:我们对深海微生物生态功能的理解,可能长期受到采样方式偏差的干扰。传统Niskin瓶等采样方式所得到的数据,并不能完全代表深海微生物的真实状态。
接下来,研究团队从多个维度评估样本的差异性,包括原核与真核微生物的表达丰度与多样性差异、保守基因表达水平(如40个单拷贝核心基因)、关键功能通路活跃度(如碳、氮、硫代谢)等。结果表明,采用MISNAC和ISMIFF方式所获得的样本具有更高和更相似的生物多样性与群落结构,更准确地反映深海微生物的原位生态功能。相比之下,Niskin采水瓶采样过程中,深海微生物因压力和温度变化导致温度敏感的异养菌交替假单胞菌快速生长,群落结构和基因表达的显著改变影响了环境适应机制、代谢途径和群落结构分析的准确性。原位样本还揭示了深海微生物的关键代谢通路基因的高活性表达,这些功能在Niskin样本中被严重低估。例如,MISNAC和ISMIFF中广泛检测到CO2固定(rbcL)、一氧化碳氧化(cox基因)、甲烷氧化(pmo基因)、氨氧化(amo基因)、硫代谢(apr基因)保持较高表达,显著高于它们在ISMIFU和Niskin样品中的表达量。 这些结果暗示了深海自养型微生物更活跃,能比以往研究结果更高效地利用无机碳生产有机质,从而支撑着深海生态系统。
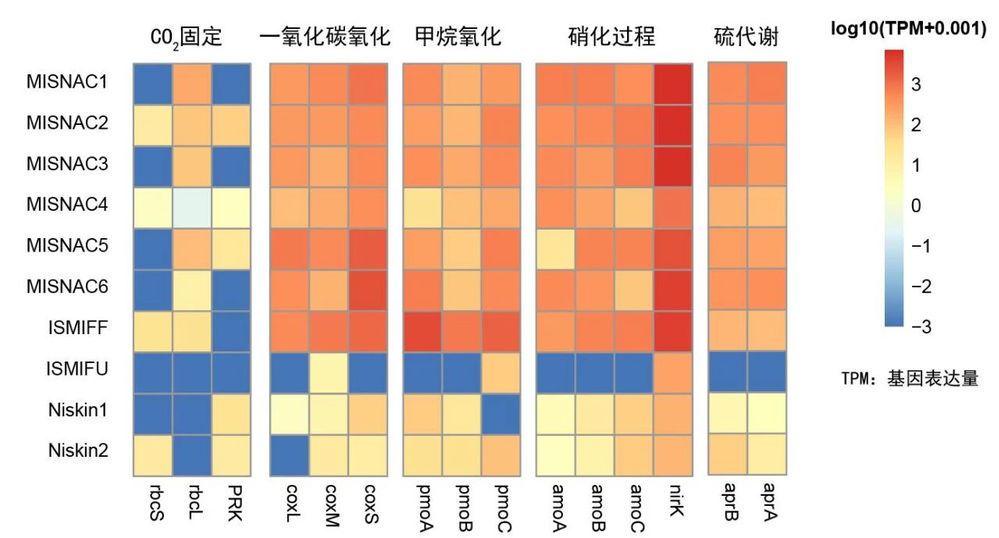
不同采样方式获取的功能基因的转录表达变异
研究提出,未来利用深海微生物功能基因研究碳、氮、硫循环、建模乃至开发新型海洋功能酶时,应采用MISNAC或ISMIFF等具备原位保真能力的技术手段。原位采样将不仅是“工程学创新”,而且是深海生态学构建基础数据的必备方法。此外,这项研究还为海洋环境保护、深海碳汇评估、极端生境功能微生物资源发掘等提供了新思路。例如,准确监测CO氧化、甲烷氧化等通路活性,可帮助我们量化深海生态系统在全球碳平衡中的真实贡献;而识别原位活跃的低丰度类群,也可能帮助提供新的微生物资源。文中的结果也暗示其他寒冷、黑暗和无氧水体中微生物采样方式也应该重视原位取样手段的应用。
近日,相关成果以为“不同采样方法下的深海微生物转录差异比较”(Transcriptional Difference of Deep-Sea Microorganisms under Different Sampling Methods)为题在线发表在《环境科学与技术》(Environmental Science & Technology)上。清华大学深圳国际研究生院2023级博士生何颖慧为本论文的第一作者,王勇为通讯作者。本研究得到了国家自然科学基金、深圳市海洋生态前沿技术重点实验室等项目的支持。
论文链接:https://pubs.acs.org/doi/10.1021/acs.est.4c05624
文:何颖慧
编辑:彭锦涛
审核:王勇


